Investigadors de la Universitat Pompeu Fabra, Institut Hospital del Mar d’Investigacions Mèdiques (IMIM) i la Universitat de Vic han desenvolupat un lloc web per estudiar l’entorn molecular de les proteïnes implicades en les malalties. El lloc web és una eina prometedora per estudiar els mecanismes moleculars subjacents a una malaltia, per entendre les relacions entre dues malalties i proposar possibles medicaments per al seu tractament.
Quim Aguirre, l’estudiant de doctorat que està duent a terme aquest projecte, ens ho explica.
Una xarxa de proteïnes interactuant entre elles
El genoma humà està format per molècules que defineixen les característiques de cada ésser humà: els gens. Els gens codifiquen per a les proteïnes, les molècules que executen les funcions específiques per mantenir el funcionament correcte del nostre cos. Les proteïnes interactuen entre elles i amb altres biomolècules per completar amb èxit les seves funcions. Des d’una perspectiva global, cada cèl·lula del cos humà pot ser vista com una xarxa complexa de proteïnes que interactuen específicament amb altres proteïnes per executar les funcions de la cèl·lula. Això es coneix com a xarxa d’interacció de proteïnes o més recentment com a interactoma humà.
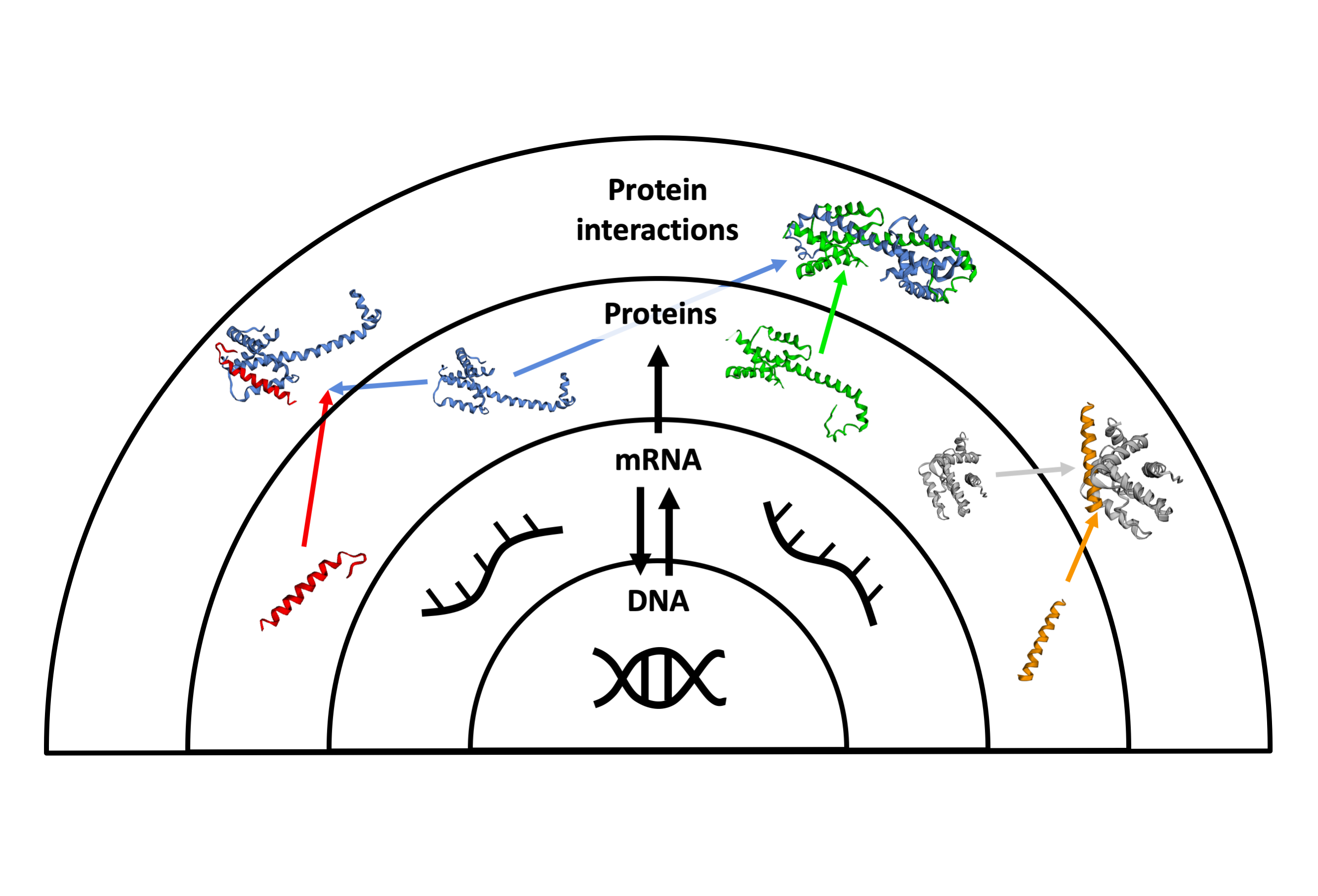
Malalties: una història d’interaccions errònees
No obstant això, el funcionament de les proteïnes no sempre és correcte. Les alteracions de gens, també anomenades mutacions, en alguns casos condueixen a un comportament inusual de les proteïnes i donen lloc a malalties. A més, les malalties complexes com el càncer, la diabetis, els trastorns neurodegeneratius o les malalties cardiovasculars comporten múltiples mutacions, cosa que complica l’estudi dels seus mecanismes moleculars. Per tant, l’estudi de la xarxa d’interacció de proteïnes és rellevant per entendre completament els mecanismes moleculars que causen la malaltia i trobar potencials tractaments.
Estudiar la xarxa d’interacció de proteïnes és esencial per a entendre els mecanismes moleculars que causen les malalties i a trobar tractaments
A la recerca de les interaccions subjacents a les malalties
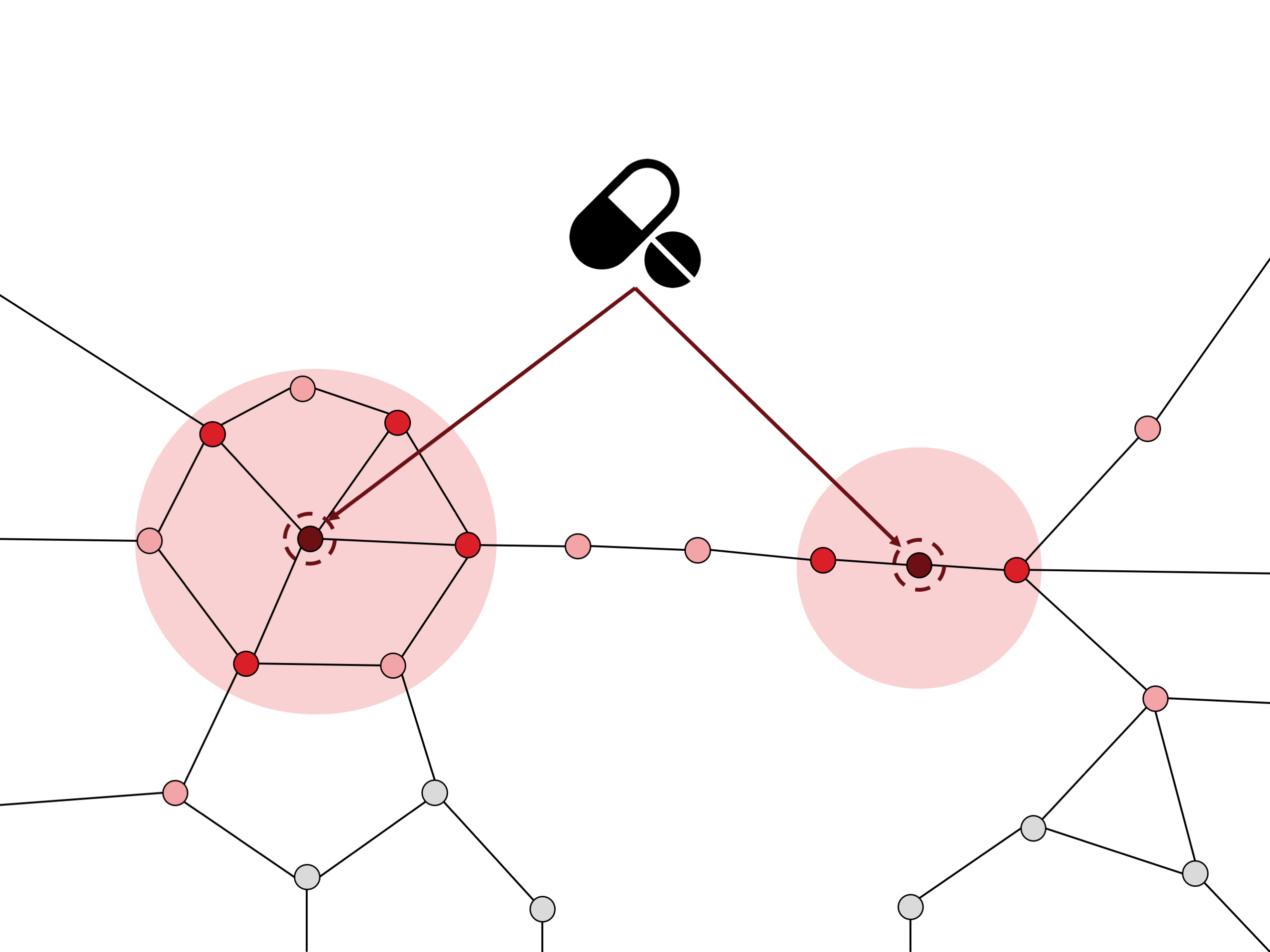
El lloc web GUILDify ha estat desenvolupat al Grup de Bioinformàtica Estructural, liderat per Baldo Oliva, amb l’objectiu d’estudiar les interaccions de proteïnes implicades en una malaltia. En accedir al lloc web, l’usuari pot introduir el nom de qualsevol malaltia i les espècies de l’interactoma a estudiar (humà, ratolí, rata …). Després d’introduir el nom de la malaltia, GUILDify proporciona a l’usuari una llista de proteïnes que probablement estan mutades en aquesta malaltia. Les mutacions s’obtenen de DisGeNET, una base de dades de gens associats a malalties humanes. GUILDify utilitza aquestes proteïnes com a llavors per a un algorisme que cerca les proteïnes de l’interactoma que estan més connectades a elles. Al final, l’usuari obté les interaccions de proteïnes que tenen més probabilitats de causar la malaltia i una llista de les funcions biològiques afectades.
GUILDify utilitza les proteïnes que se sap que estan mutades en una malaltia com a llavors per a un algorisme que cerca altres proteïnes de l’interactoma que estan connectades a elles
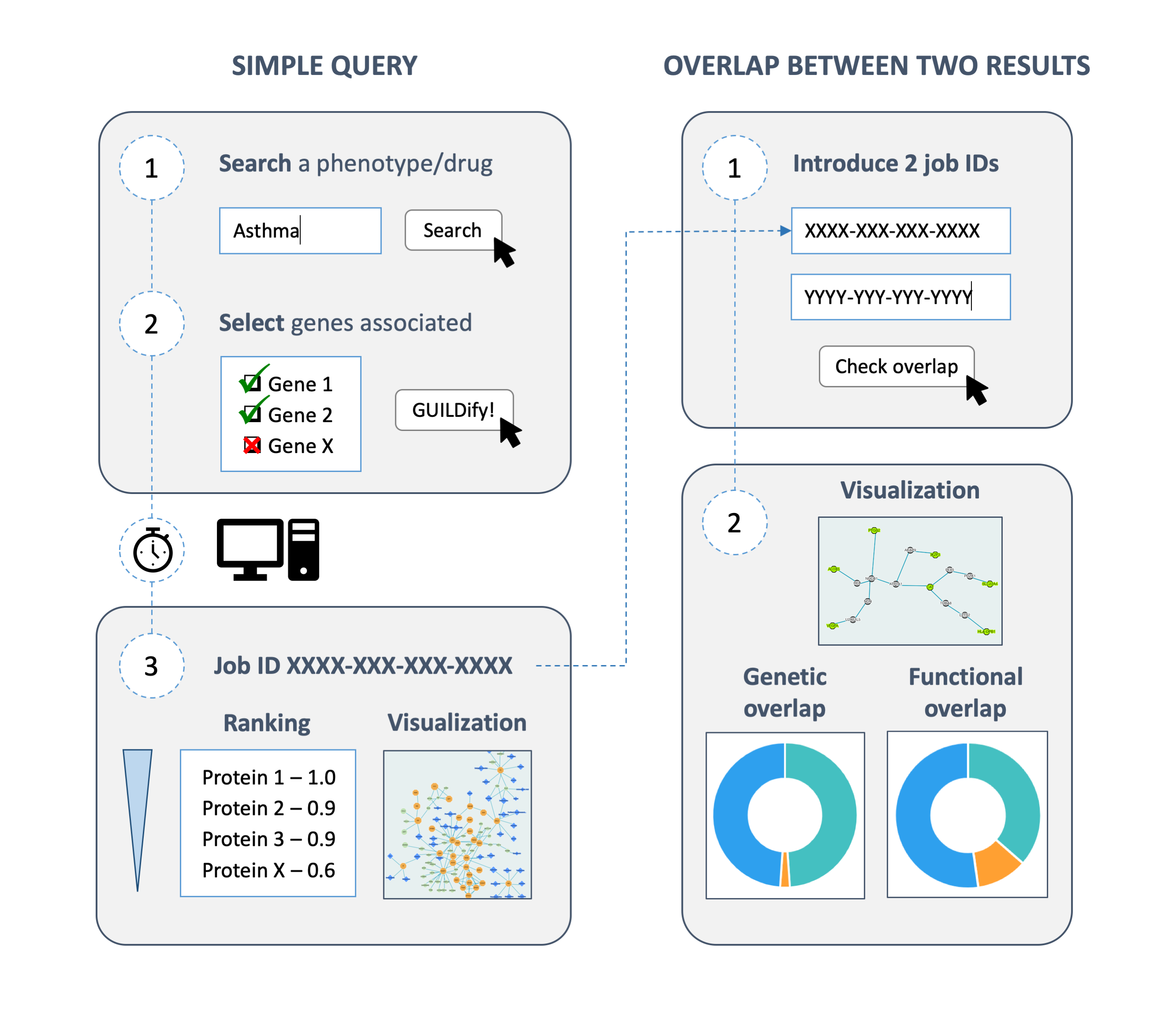
A més de l’estudi de malalties de forma individualitzada, GUILDify també s’ha adaptat per estudiar la relació entre dues malalties. Hi ha parelles de malalties denominades comorbilitats que tenen més probabilitats de produir-se simultàniament en un mateix pacient. En alguns casos, les comorbilitats es poden explicar perquè comparteixen un mecanisme molecular comú. En un estudi previ de Carlota Rubio-Pérez i companys del Departament de Ciències Experimentals i de la Salut, Universitat Pompeu Fabra (DCEXS-UPF) i de l’IMIM, el programari de GUILDify es va utilitzar per predir les comorbiditats amb èxit. Motivat per aquesta recerca, el lloc web GUILDify inclou l’opció d’introduir dues malalties i explorar les proteïnes i funcions biològiques implicades comunes a ambdues.
De la malaltia al tractament
GUILDify també ofereix l’opció d’estudiar els mecanismes moleculars dels fàrmacs. En introduir el nom d’un medicament, GUILDify proporciona a l’usuari una llista de proteïnes diana amb les quals el fàrmac interacciona per produir un efecte terapèutic. De la mateixa manera que fa amb les malalties, el programa cerca les proteïnes de l’interactoma que estan més connectades amb les proteïnes diana del fàrmac. Les proteïnes que GUILDify retorna són les que probablement rebran un major impacte del fàrmac. Per tant, es pot utilitzar GUILDify per veure si la llista de proteïnes afectades per una malaltia és similar a la llista de proteïnes afectades per l’acció d’un medicament. Aquest és un indicador de l’impacte que té el fàrmac en l’entorn molecular de la malaltia.

Quim Aguirre Plans és llicenciat en Biotecnologia per la Universitat Autònoma de Barcelona (UAB) i té un màster en Bioinformàtica per la Universitat Pompeu Fabra (UPF). Ara està immers en el seu doctorat en el Grup de bioinformàtica estructural (DCEXS-UPF), on estudia els efectes terapèutics i tòxics dels medicaments a la xarxa d'interaccions de proteïnes.