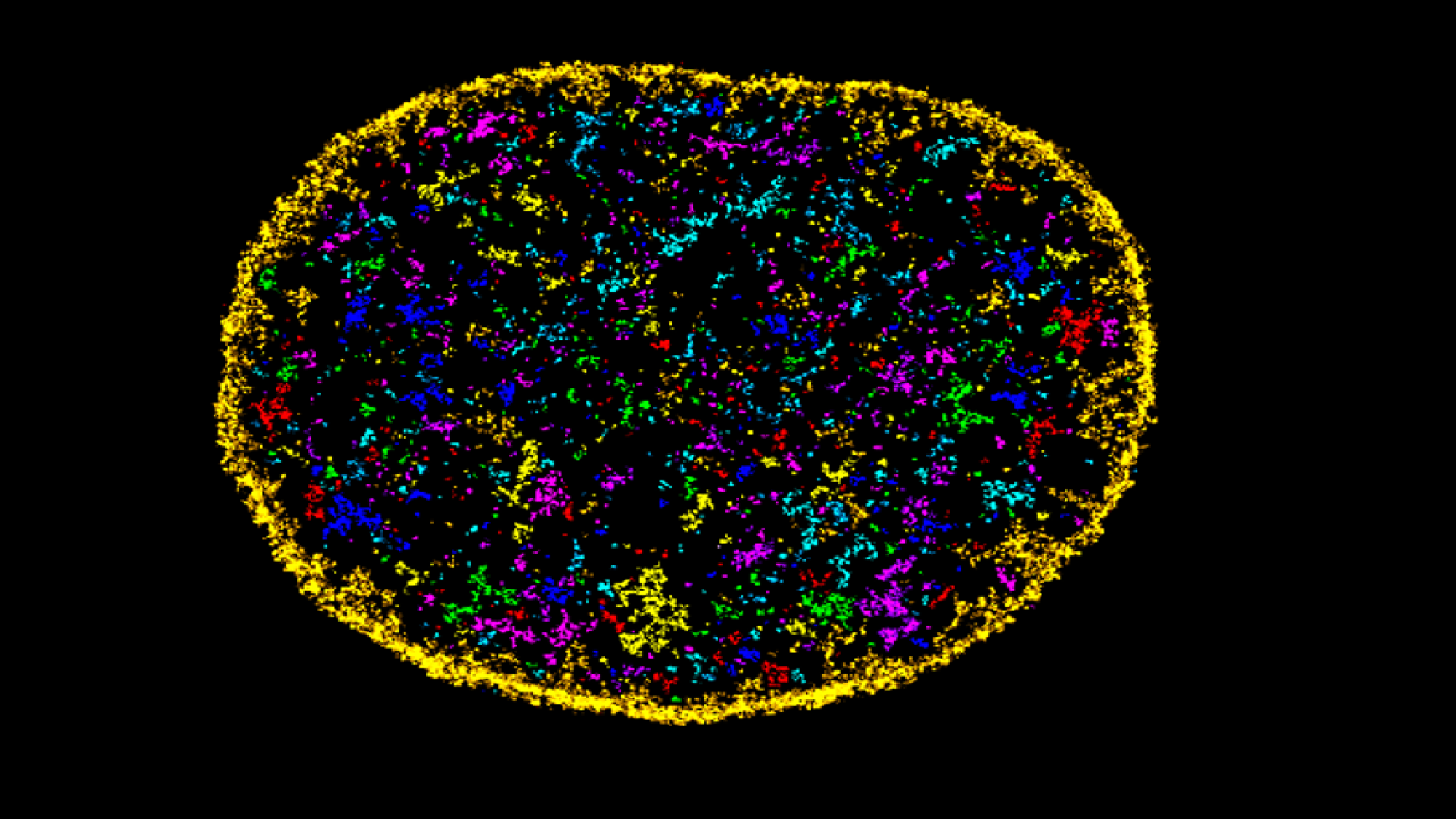
Equipos investigadores del Centro de Regulación Genómica (CRG) y de la Universidad del País Vasco han desarrollado una herramienta de inteligencia artificial, AINU (IA del NÚcleo), capaz de escanear imágenes de células a muy alta resolución, a un nivel de precisión nanométrico, y detectar reordenamientos de tan sólo 20 nm, es decir, 5.000 veces menos que la anchura de un cabello humano.
«Estos reordenamientos pueden ser cambios en la organización del ADN dentro de las células o en la distribución de enzimas en el núcleo, ambas cosas pueden cambiar en las células cancerosas. O pueden ser ligeras diferencias en la densidad del ADN tras una infección vírica», explica Pia Cosma, profesora de investigación ICREA, jefa de grupo en el CRG y responsable del estudio.
De hecho, la IA fue capaz de detectar cambios en el núcleo de una célula tan pronto como una hora después de ser infectada por el virus del herpes simple tipo 1.
Las aplicaciones clínicas de esta herramienta están aún a años vista, porque precisa de imágenes tomadas con una técnica especial de microscopía llamada STORM, que sólo puede analizar unas pocas células a la vez. Además, se trata de un equipo muy especializado que requeriría grandes inversiones.
Pero el equipo investigador espera que AINU sea ya útil en la investigación científica, por ejemplo identificando células madre con una precisión muy alta, lo que ayudaría a que las terapias con células madre fueran más seguras y eficaces.
Un algoritmo detectivesco para seleccionar el mejor fármaco
Otra herramienta, RTDetective, ha sido desarrollada conjuntamente por el CRG y el IRB. RTDetective es capaz de predecir qué fármacos serán más eficaces para tratar enfermedades causadas por «mutaciones sin sentido», un tipo de mutaciones que actúan como una señal de stop que impide la síntesis de la proteína completa.
Ya existen «terapias de supresión de mutaciones sin sentido», fármacos que ayudan a las células a ignorar estas señales de parada y continuar con la producción de proteínas. Pero no todos funcionan igual de bien para todas las mutaciones, porque su capacidad para permitir que la maquinaria celular «lea» la mutación sin sentido depende no sólo de la propia mutación, sino también del código genético que la rodea inmediatamente.
«A un paciente se le diagnostica un trastorno genético, se identifica la mutación mediante pruebas genéticas y un modelo informático sugiere el mejor fármaco. Este tipo de toma de decisiones informada es la promesa de la medicina personalizada que esperamos desbloquear en el futuro».
Ben Lehner, Profesor de Investigación ICREA, CRG e Instituto Wellcome Sanger
El equipo investigador analizó 5.800 señales de parada prematura causantes de enfermedades y probó la eficacia de ocho fármacos distintos en cada una de ellas. Los datos (140.000 mediciones individuales) fueron lo suficientemente amplios como para entrenar modelos predictivos precisos, que se utilizaron para crear RTDetective.
El siguiente objetivo de la investigación es confirmar la funcionalidad de las proteínas producidas mediante estos fármacos, un paso crucial para validar su aplicabilidad clínica.
Carnevali, D., Zhong, L., González-Almela, E. et al. A deep learning method that identifies cellular heterogeneity using nanoscale nuclear features. Nat Mach Intell (2024). https://doi.org/10.1038/s42256-024-00883-x
Toledano, I., Supek, F. & Lehner, B. Genome-scale quantification and prediction of pathogenic stop codon readthrough by small molecules. Nat Genet (2024). https://doi.org/10.1038/s41588-024-01878-5