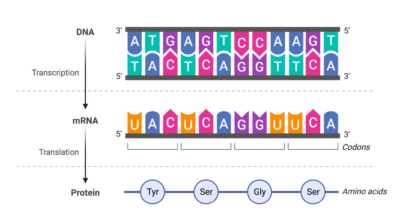
Todos los seres vivos fabrican proteínas a partir de su ADN, pero no todas se conocen. De algunas no se sabe su forma ni su función, pero sí se puede tener la secuencia de ADN que las codifica. Esta secuencia ha sido la base de una colaboración entre el Centro Andaluz de Biología del Desarrollo (CABD) y el Instituto de Biología Evolutiva (IBE: CSIC-UPF) para analizar proteínas con inteligencia artificial.
El estudio ha analizado secuencias de organismos modelo (levadura, ratón y mosca de la fruta) a través del deep learning o aprendizaje profundo, y se han podido determinar y clasificar con gran detalle las funciones de proteínas de las que no se tenía información previa.
A través del aprendizaje profundo, los grupos de investigación han logrado determinar la función de proteínas de las que solo se tenía la secuencia de ADN que las codifica.
Los y las autoras también observaron que, de entre los dos métodos de deep learning utilizados, los modelos de lenguaje o transformers son más eficientes que las redes convolucionales. Este último método se basa en el procesamiento de imágenes, mientras que los transformers procesan secuencias y lenguaje, lo que los hace más informativos y precisos, además de poder recuperar información a partir de secuencias de ARN.
Esta investigación es vital para abordar la problemática del proteoma oscuro, formado por todas aquellas proteínas de las que no se tiene información. Así, se pueden analizar proteínas e identificar funciones de genes con potencial biomédico y biotecnológico, sobre todo en organismos poco estudiados hasta ahora, señala Rosa Fernández, co-líder del estudio en el IBE (CSIC-UPF). Esto es especialmente relevante ahora que se están secuenciando grandes cantidades de organismos desconocidos, lo que genera millones de secuencias, de las cuales no se puede predecir la función usando métodos tradicionales.
Israel Barrios-Núñez, Gemma I Martínez-Redondo, Patricia Medina-Burgos, Ildefonso Cases, Rosa Fernández, Ana M Rojas, Decoding functional proteome information in model organisms using protein language models, NAR Genomics and Bioinformatics, Volume 6, Issue 3, September 2024, lqae078, https://doi.org/10.1093/nargab/lqae078