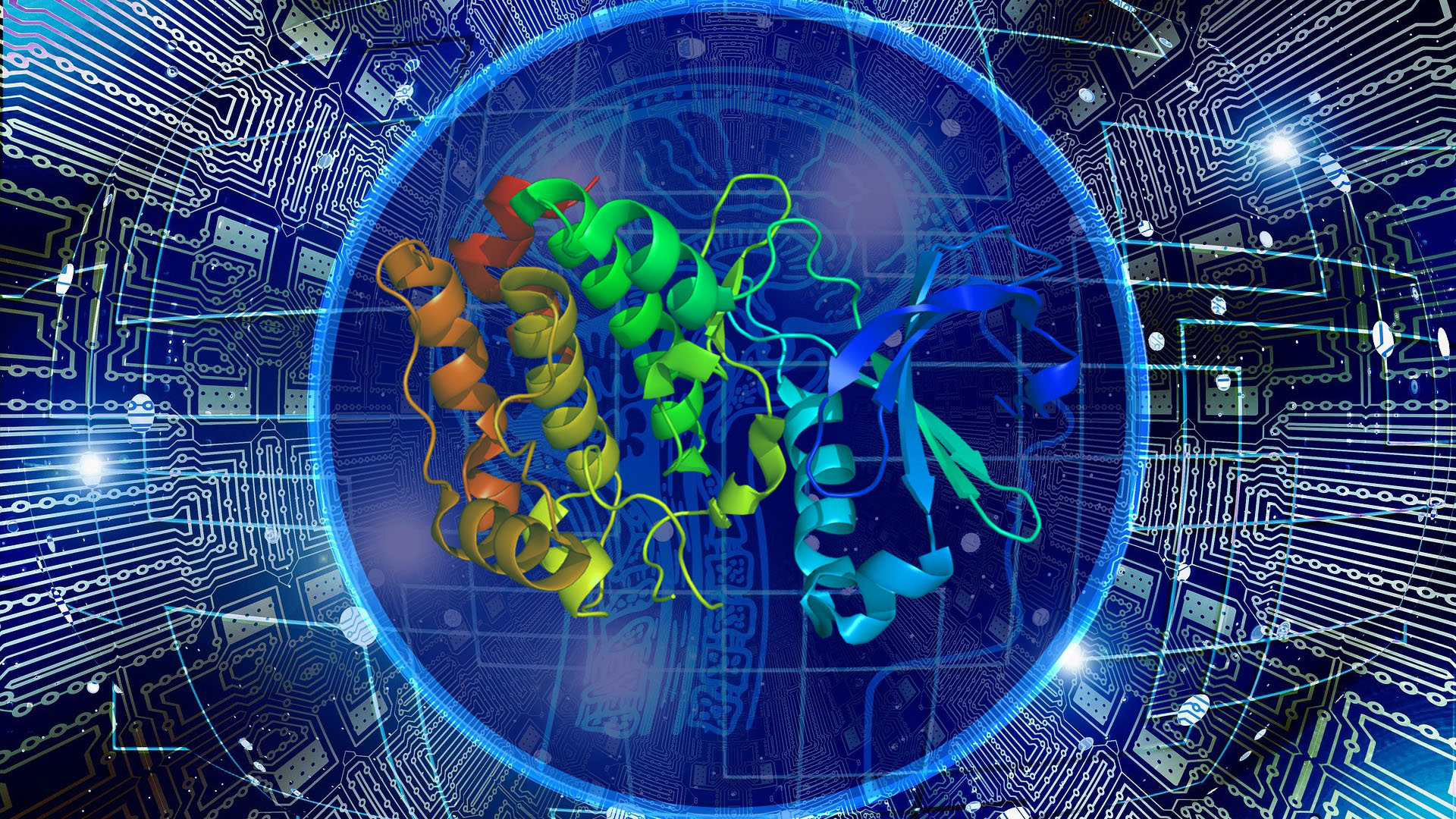
Conèixer l’estructura de les proteïnes – els blocs elementals que conformen les cèl·lules – és essencial per comprendre la seva funció, i per tant per entendre com funcionen els organismes i com i per què tenen lloc les malalties.
Hi ha dues formes principals per descobrir l’estructura d’una proteïna:
- Experimental: a través de cristal·lografia, ressonància magnètica, etc.
- Teòrica: amb ordinadors, a través de la comparació de seqüències amb proteïnes conegudes, etc.
Durant més de 25 anys, el concurs internacional bianual CASP (Critical Assessment of protein Structure Prediction) ha ajuntat a experimentalistes i investigadors teòrics per a posar a prova els models predictius d’aquests últims.
Fa només uns anys, encertar el 10% de les estructures proteiques proposades es considerava un èxit, i fa dos anys l’equip de Google va sorprendre la comunitat científica en encertar un 50%. En el més recent CASP, el resultat del qual es va anunciar a la fi del 2020, la companyia DeepMind – pertanyent a Google – va utilitzar el seu sistema d‘intel·ligència artificial AlphaFold i va aconseguir una predicció correcta del 100%, per a gran sorpresa de tots els i les participants, causant una revolució en aquest camp.
En l’últim episodi del podcast dirigit al públic general, “Peter Parking”, Baldo Oliva, líder del grup de bioinformàtica estructural del GRIB, al Parc de Recerca Biomèdica de Barcelona (PRBB), ens explica com la intel·ligència artificial (i específicament el deep learning) acaba de transformar la predicció de l’estructura proteica.