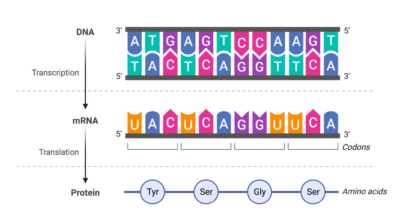
Todas y cada una de las proteínas de nuestro cuerpo están constituidas por cadenas de aminoácidos. Cuando uno de estos aminoácidos sufre una mutación, toda la proteína puede verse afectada, a menudo marcando la diferencia entre salud y enfermedad. Esto ocurre porque la estructura de la proteína cambia, lo que puede impedir su función. Pero medir experimentalmente cómo diferentes mutaciones afectan la estructura y función de una proteína resulta imposible, ya que hay demasiadas combinaciones a probar. Por poner un ejemplo, en una proteína formada por 34 aminoácidos, existen 17 mil millones de combinaciones diferentes si solo se permite un único cambio en cada posición. Y la mayoría de las proteínas humanas son mucho más grandes que esto.
Hasta hace poco, se asumía que a menudo una mutación podía afectar a otra, aumentando o suprimiendo sus efectos. Sin embargo, un estudio del Centro de Regulación Genómica (CRG), en colaboración con el Wellcome Sanger Institute, ha descubierto que las interacciones entre mutaciones son menos comunes de lo que se creía. Esto significa que la gran mayoría de mutaciones afectan a una proteína de manera independiente.
Descubrir que las mutaciones no suelen interactuar entre ellas significa que la estabilidad de las proteínas se ve afectada por las mutaciones siguiendo reglas mucho más simples que los cálculos que se hacían hasta ahora. El equipo de investigación, dirigido por André Faure y Ben Lehner, analizó secuencias de proteínas con diferentes combinaciones de mutaciones y probó su estabilidad. El resultado experimental fue muy similar al de los modelos que calculaban el efecto total de las mutaciones como una simple suma de todas ellas, es decir, considerándolas independientes. Sin embargo, hay que tener en cuenta que el modelo fue capaz de predecir la estructura de la proteína si esta tenía como máximo dos mutaciones.
«Hemos visto que no necesitamos superordenadores para predecir el comportamiento de una proteína; solo hacen falta algunas medidas y matemáticas sencillas.»
Ben Lehner (CRG y Wellcome Sanger Institute)
Aun así, todavía se requiere un cierto nivel de validación experimental para confirmar las predicciones, especialmente en aplicaciones críticas como el desarrollo de fármacos, donde pueden existir efectos imprevistos o interacciones poco frecuentes que los modelos no capturan. Pero esta investigación ayudará a optimizar el número de experimentos necesarios para analizar la estructura de las proteínas y poderla predecir, lo que supone un avance en el diseño de proteínas con potencial farmacológico y biotecnológico.
Faure, A.J., Martí-Aranda, A., Hidalgo-Carcedo, C. et al. The genetic architecture of protein stability. Nature (2024). https://doi.org/10.1038/s41586-024-07966-0