Este texto ha sido escrito por Ivan Milenkovic, estudiante de doctorado en el laboratorio dirigido por Eva Novoa en del Centro de Regulación Genómica (CRG). Ivan ha ganado el primer premio del concurso de divulgación científica Rin4′ con su presentación “La vida dinàmica dels ribosomes”. En este artículo nos cuenta más sobre su investigación sobre los ribosomas, unos orgánulos celulares encargados de la formación de las proteínas y que, hasta hace relativamente poco, se creía que eran estáticos y uniformes en todos los tejidos. Ivan también comparte su experiencia como estudiante de doctorado hasta ahora.
También puedes leer una entrevista que le hicieron desde la UPF aquí.
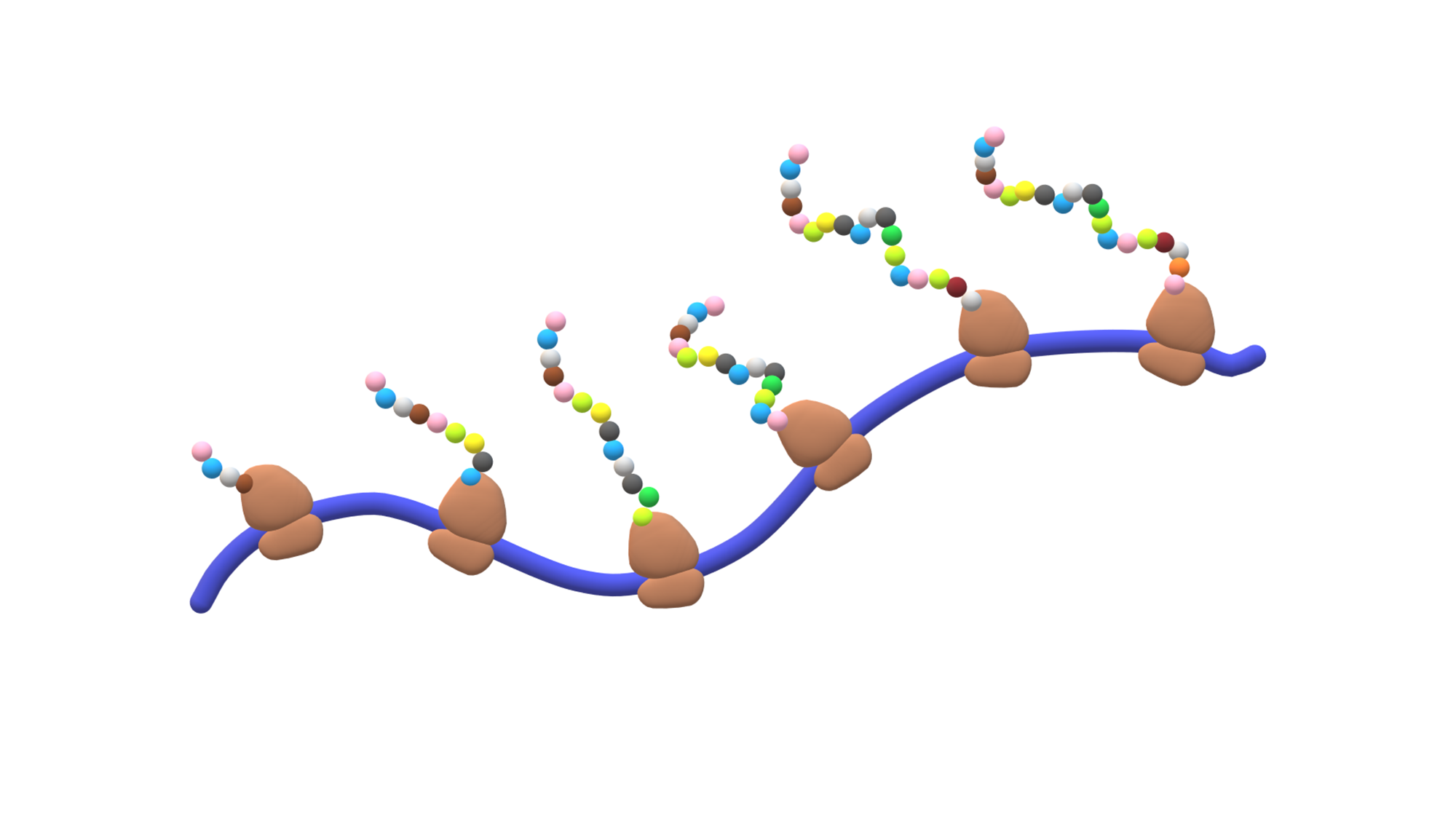
El objetivo de mi proyecto es comprender el papel biológico de la heterogeneidad de los ribosomas. Durante mucho tiempo hemos pensado que los ribosomas – las máquinas moleculares responsables de la síntesis de proteínas en todos los seres vivos – tienden a ser uniformes, con una composición estática que es idéntica en todos los tejidos de nuestro cuerpo. Sin embargo, recientemente se ha descubierto que la estructura de los ribosomas puede ser mucho más heterogénea, y que esta heterogeneidad puede ser específica de cada tejido o etapa de desarrollo. En mi tesis doctoral, trato de entender si esta diversidad estructural comporta una función especializada.
Los ribosomas son las máquinas moleculares responsables de la síntesis de proteínas en todos los seres vivos.
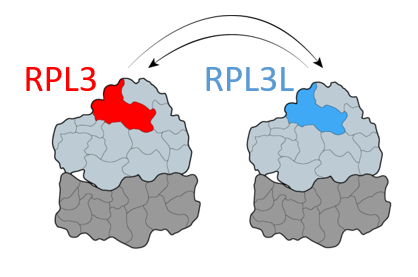
Algunas proteínas ribosómicas tienen parálogos, es decir, proteínas que provienen de un gen ancestral que se duplicó durante la evolución. Hemos descubierto que una proteína ribosomal llamada RPL3L solo se expresa y se incorpora a los ribosomas en los músculos estriados, a diferencia de su paráloga, RPL3, que se expresa en todos los demás tejidos. Al generar un modelo de ratón knockout de Rpl3l (un ratón que no tiene el gen Rpl3l), hemos identificado que en ausencia de RPL3L, RPL3 se expresa también en los músculos estriados y compensa la pérdida de su parálogo. Además, hemos encontrado que las interacciones ribosoma-mitocondria cambian dependiendo de qué proteína se incorpore al ribosoma, lo que sugiere que la elección del parálogo de la proteína ribosómica puede afectar a la función del ribosoma.
La heterogeneidad del ribosoma también puede provenir del ARNr, especialmente de las modificaciones del ARNr. En el laboratorio, hemos desarrollado un método para estudiar las modificaciones del ARN mediante la secuenciación de moléculas de ARN nativas utilizando la plataforma tecnológica Oxford Nanopore. Hemos secuenciado el ARNr de varios tejidos de ratón en diferentes etapas de desarrollo y hemos descubierto que varios nucleótidos se modifican de forma dinámica, tanto a nivel de los tejidos como de las etapas de desarrollo.
En conjunto, estos resultados coinciden con varios estudios de los últimos años que describen a los ribosomas como mucho más dinámicos de lo que se pensaba.
Ordenadores y poyata, un reto apasionante
Actualmente estoy en el cuarto año de mi doctorado. El principal reto hasta ahora ha sido aprender bioinformática mientras hacía experimentos al mismo tiempo. Al tener una formación en bioquímica, casi no tenía experiencia en bioinformática cuando empecé mi doctorado. Sin embargo, mi supervisora, Eva Novoa, anima encarecidamente a todos y todas las estudiantes de doctorado a participar tanto en el laboratorio experimental como en la parte bioinformática, así que desde mis primeros días en el laboratorio estuve inmerso en este entorno que me ayudó a trabajar en ambos en paralelo. Esto ha sido lo más gratificante, ya que disfruto mucho participando en todas las etapas del proyecto: ¡desde los primeros diseños experimentales hasta los últimos análisis bioinformáticos y la visualización!