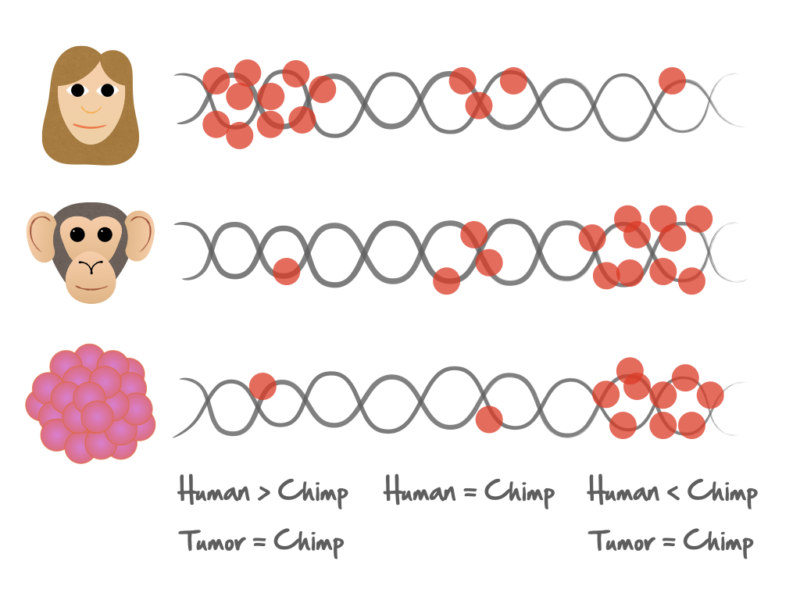
A partir de los datos del proyecto PanCancer, un equipo de investigación del Instituto de Biología Evolutiva (IBE: CSIC-UPF) ha mirado en qué regiones del genoma se acumulan más mutaciones, comparando el genoma de humanos, de chimpancés y gorilas, y de células humanas cancerosas. Sorprendentemente, han visto que la distribución de mutaciones en muestras humanas de cáncer es más parecida a la de los grandes simios sanos que a la de humanos sanos.
¿La razón?
Parece que la compleja historia evolutiva de los humanos – con varios ‘cuellos de botella’ donde la población humana ha disminuido drásticamente – ha “borrado” estas señales de acumulación de mutaciones que deberíamos tener. Es decir, en esas fases de alta mortalidad humana, al desaparecer parte importante de la población, disminuye la variabilidad general. Y esto afecta sobretodo a las regiones donde había más variabilidad (o sea, más mutaciones). Esto no ha ocurrido en chimpancés o gorilas, cuyas poblaciones no han sufrido estos cuellos de botella.
La compleja historia evolutiva de los humanos ha “borrado” la acumulación de mutaciones que suele ocurrir en los tumores – esto no ha ocurrido en los grandes simios, que las mantienen.
¿Y qué implica esto?
Estos resultados, publicados en acceso abierto, explicarían porqué las mutaciones en células tumorales y las mutaciones que se dan normalmente en humanos se concentran en zonas muy distintas del genoma. No sería, como se creía hasta ahora, por un comportamiento especial de las células cancerosas, sino por la historia evolutiva humana. Así, según Txema Heredia, primer autor del estudio dirigido por Arcadi Navarro, para entender cómo se acumulan las mutaciones en las células humanas y comprender mejor los tumores, seria más útil mirar cómo se acumulan en grandes simios en lugar de hacerlo en humanos.
Heredia-Genestar JM; Marquès-Bonet T; Juan D; Navarro A. Extreme differences between human germline and tumor mutation densities are driven by ancestral human-specific deviations. Nature Communications, May 2020. DOI: 10.1038/s41467-020-16296-4.