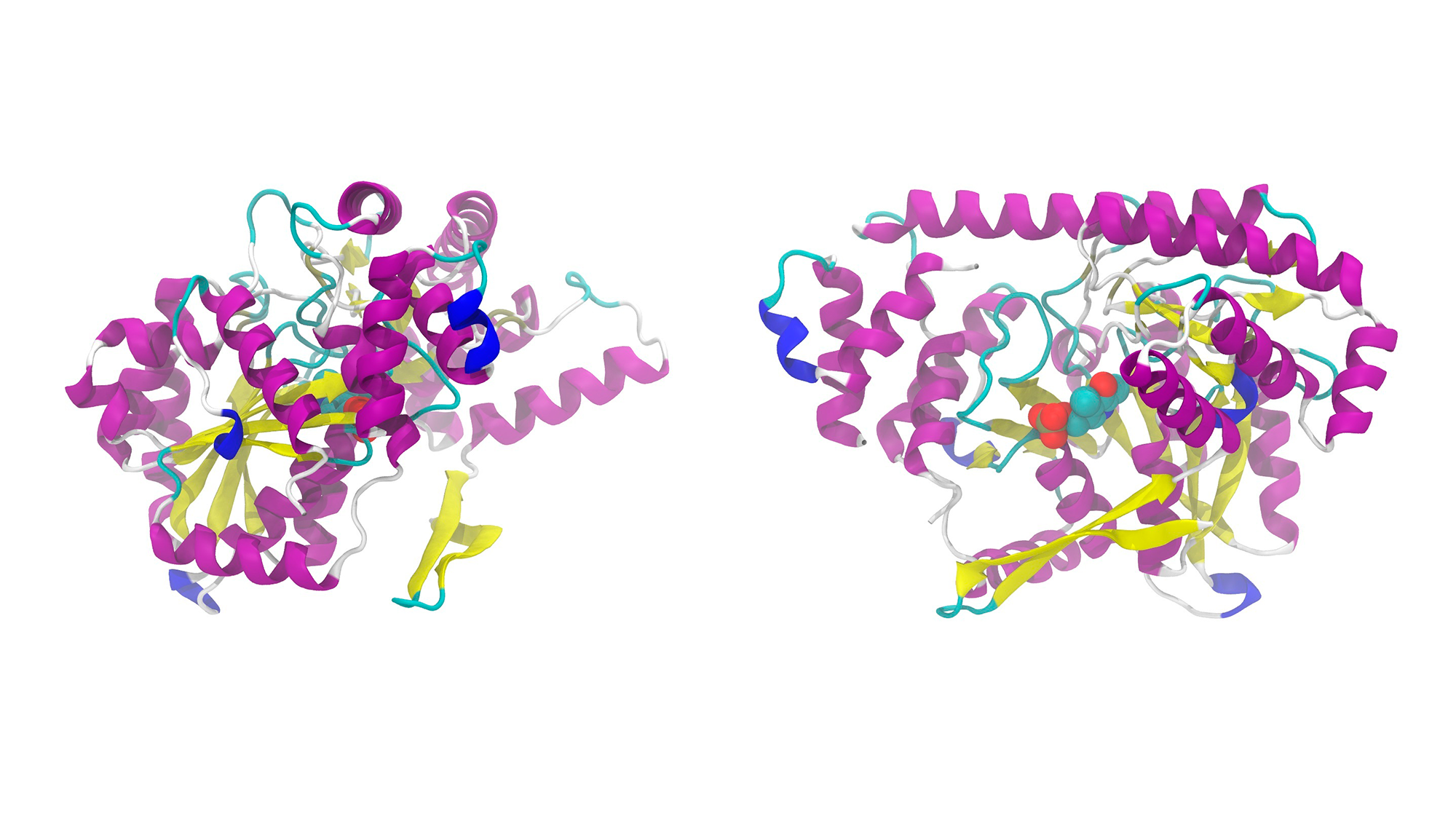
Un estudi portat a terme entre el Centre de Regulació Genòmica (CRG) i l’empresa BGI de Shenzen ha determinat que les mutacions amb canvi de sentit a l’ADN que causen malalties ho fan majoritàriament perquè desestabilitzen la proteïna que codifiquen. L’equip ho va observar en un 60% de mutacions que van estudiar – introduint-les en cèl·lules de llevat – i es deu a que la mutació resulta en un pitjor plegament de la proteïna codificada.
No obstant això, l’equip dirigit per Ben Lehner també va observar vàries mutacions que no desestabilitzen les proteïnes, però que també causen malalties. Això obre la porta a nous tractaments segons les conseqüències de la mutació. Algunes de les malalties que van estudiar són les cataractes hereditàries, la miopatia amb cossos reductors, el síndrome de Hay-Wells o el de Rett.
Aquest estudi ha estat possible gràcies al projecte Dominioma Humà 1 que ha catalogat més de mig milió de mutacions en al voltant de 500 dominis proteics humans. Aquestes mutacions van ser creades canviant sistemàticament cada aminoàcid per tots els altres aminoàcids possibles. Tot i que és el catàleg d’aquest estil més gran fins al moment, només cobreix el 2,5% de les proteïnes humanes que es coneixen.
De les malalties a l’arbre de la vida
En una altra investigació del CRG, dirigida per Cedric Notredame, es van combinar les dades sobre l’estructura de les proteïnes amb les de les seqüències genòmiques per a determinar millor els arbres filogenètics. Aquests són clau per a entendre l’evolució de la vida a la terra, així com controlar la dels patògens o desenvolupar nous medicaments.
Tradicionalment, els arbres filogenètics han estat creats només amb dades de les seqüències d’ADN o de proteïnes i mirant les semblances i les diferències entre els ancestres. Per desgràcia, els canvis entre seqüències d’ADN al llarg del temps poden ser tan grans que es perd la línia que uneix les formes ancestrals amb les modernes.
No obstant això, tot i les mutacions, l’estructura de les proteïnes es manté bastant constant. Aquí, s’ha combinat l’estudi dels canvis de l’estructura predita computacionalment, especialment la distància entre els aminoàcids, amb els canvis de seqüència d’ADN per a dibuixar més precisament els arbres evolutius. També ha estat necessària la intel·ligència artificial que ha predit l’estructura de les proteïnes, així com ha aportat dades fins ara desconegudes.
Beltran, A., Jiang, X., Shen, Y. et al. Site-saturation mutagenesis of 500 human protein domains. Nature 637, 885–894 (2025). https://doi.org/10.1038/s41586-024-08370-4
Baltzis, A., Santus, L., Langer, B.E. et al. multistrap: boosting phylogenetic analyses with structural information. Nat Commun 16, 293 (2025). https://doi.org/10.1038/s41467-024-55264-0