En aquests moments on l’aparició del SARS-CoV-2 ha canviat les nostres vides, és un bon moment per reflexionar sobre com aquesta pandèmia també afecta a la resta d’éssers vius. Per exemple, en el cas dels ximpanzés (Pan troglodytes) ja es comença a parlar de com aquesta infecció pot ser perjudicial per a la seva supervivència.
Si bé la COVID-19, i altres malalties infeccioses com l’Ebola, són un factor de risc pels ximpanzés, també la destrucció de l’hàbitat natural, el canvi climàtic i el tràfic il·legal han provocat que avui en dia els ximpanzés estiguin considerats en perill d’extinció. Segons l’IUCN (International Union for Conservation of Nature and Natural Resources), s’estima que la seva població s’haurà reduït a la meitat cap a l’any 2050. Així doncs, és imprescindible l’elaboració de plans d’acció eficaços per a la conservació d’aquesta espècie.
S’estima que la població de ximpanzés s’haurà reduït a la meitat cap a l’any 2050. La destrucció del seu hàbitat natural, el canvi climàtic i el tràfic il·legal en són les causes principals.
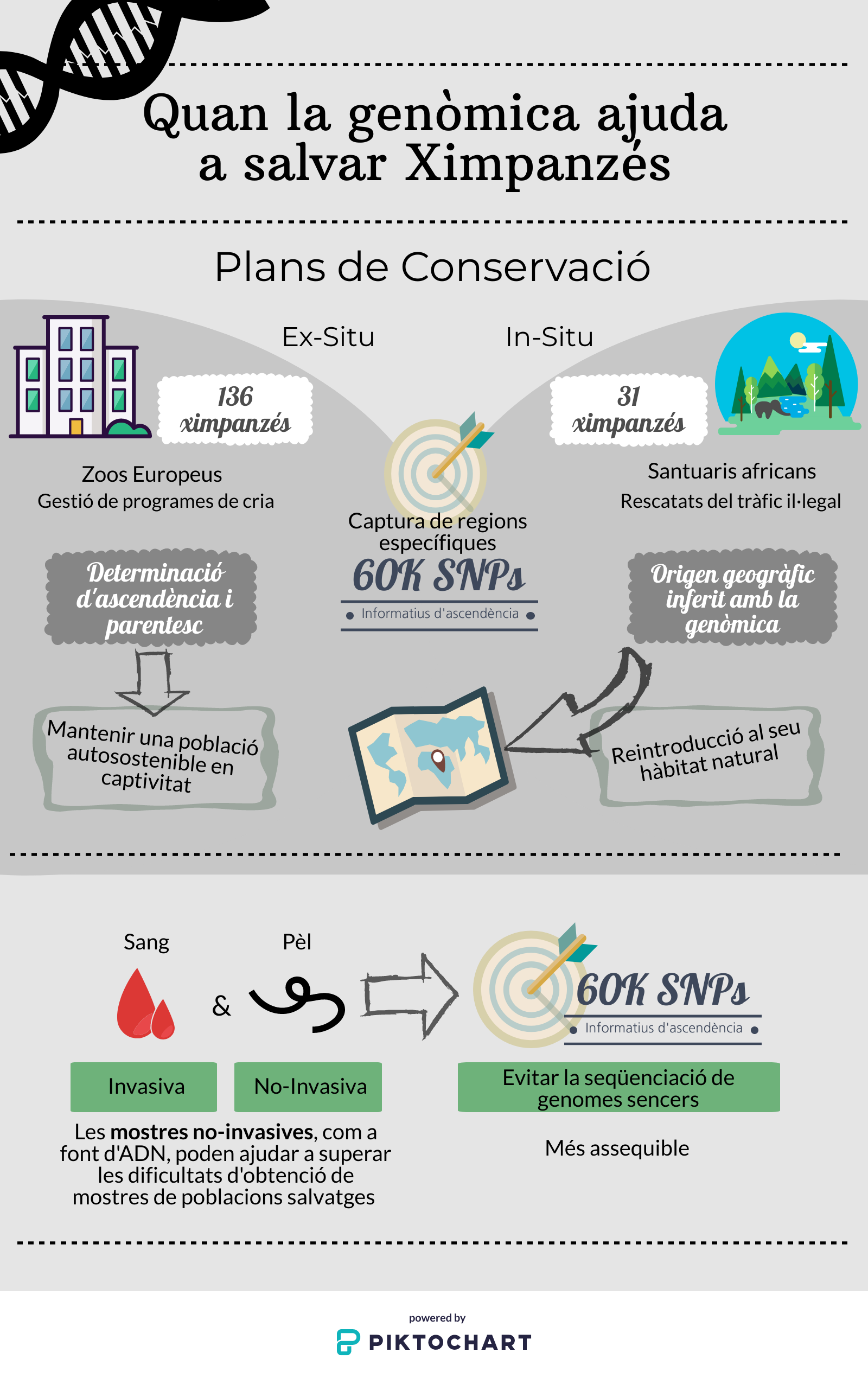
Aquests plans solen dividir-se en ex situ i in situ, depenent de si s’efectuen fora o en el seu hàbitat natural.
Fora de l’Àfrica, un dels plans de conservació d’espècies en perill d’extinció més importants és el dirigit per l’Associació de Zoos i Aquaris Europeu (EAZA). El seu objectiu principal és vetllar per la supervivència a llarg termini d’una població sana de ximpanzés, mantenint-ne separades les seves quatre subespècies. Tot i això, degut a un coneixement poc acurat de l’origen, ascendència genètica i parentesc dels ximpanzés, però també a la manca d’eines moleculars precises, la cria d’aquesta espècie ha derivat cap a casos de mescla entre subespècies i a consanguinitat.
La genòmica al rescat
Per aquest motiu, en l’estudi que hem publicat a Heredity, hem volgut explorar fins a quin punt trobem aquests casos en 167 ximpanzés tant de zoos europeus com de santuaris d’Àfrica. D’aquesta manera es poden identificar els individus susceptibles a formar part de programes de cria en captivitat i contribuir a mantenir una població genèticament fidedigna amb la natura. En l’estudi, hem determinat que dels 136 ximpanzés que formen part del programa de conservació ex situ en zoos europeus, 90 tenen ascendència d’una sola subespècie mentre que la resta han sigut classificats com a híbrids, és a dir, amb ascendència de dues o més subespècies.
Però també des d’una visió de la conservació in situ, hem completat l’estudi amb la determinació de l’origen geogràfic de 31 ximpanzés rescatats del tràfic il·legal de mascotes i que actualment es troben en santuaris a Àfrica. Això ha sigut possible gràcies a un estudi anterior del nostre grup on vam descriure un atles genètico-geogràfic que permet inferir el lloc d’origen a partir de les dades genètiques (de Manuel, 2016).
Dels 31 ximpanzés que hem analitzat, la majoria pertanyen a la subespècie P.t. schweinfurthii, i n’hem inferit l’origen principalment a dues localitzacions concretes: una al nord i l’altra al sud de la República Democràtica del Congo. Al creuar aquestes dades amb el lloc de confiscació, hem vist que en la majoria dels casos coincidia, fet que ens indica que la ruta de tràfic il·legal ocorre a escala local, a poca distància del lloc on s’ha capturat l’animal. Tot i això, també hem trobat dos casos on els ximpanzés havien estat confiscats molt més lluny, un a Moscou i l’altre a Nairobi, mentre que l’origen geogràfic inferit genèticament els situa a l’oest d’Àfrica i a Camerun, respectivament.
Determinar l’origen geogràfic dels animals confiscats obra la possibilitat que aquests ximpanzés puguin ser reintroduïts al seu hàbitat natural
El fet de poder determinar l’origen geogràfic dels animals confiscats obra la possibilitat que aquests ximpanzés puguin ser reintroduïts al seu hàbitat natural, i gràcies a les anàlisis genòmiques, poder-ho fer al lloc on toca. Però també ens permet conèixer a quines zones hi ha més casos de tràfic il·legal per a que les administracions pertinents puguin aplicar les sancions necessàries.
Solucions innovadores per les limitacions
Ara bé, molts cops l’aplicació de la genètica en la conservació s’ha vist limitada pel finançament. Per això, hem volgut desenvolupar i aplicar un mètode que ens permet capturar les regions del genoma que ens informen quina és la subespècie del ximpanzé analitzat mirant ~60.000 SNPs, tot evitant el cost de la seqüenciació de genomes complets. Així doncs, hem proposat un tècnica assequible que pot ser utilitzada en la conservació d’espècies en perill d’extinció.
Una altra limitació que molt sovint ens trobem és l’obtenció de mostres, sobretot pel que fa a animals salvatges. Per tant, també hem comprovat la idoneïtat de les mostres no-invasives (pèl), comparant els resultat amb mostres de sang dels mateixos individus.
Aquest projecte ha sigut possible gràcies a una llarga col·laboració entre el grup de Tomàs Marquès a l’Institut de Biologia Evolutiva (IBE: CSIC-UPF), amb el suport del Zoo de Barcelona, i el Zoo de Copenhaguen, amb els investigadors Peter Frandsen i Christina Hvilsom.
“El nostre estudi ajudarà a construir, amb més precisió, l’atles de correspondència genètica i geogràfica i així poder geolocalitzar ximpanzés a nivell molt més local”
Clàudia Fontserè Alemany
En definitiva, el nostre estudi demostra el potencial de la genòmica per contribuir en l’elaboració de plans de conservació però alhora ens prepara el terreny pel estudis que vindran. Els propers passos en la genètica de la conservació consistiran en construir, amb més precisió, aquests atles de correspondència genètica i geogràfica i així poder geolocalitzar ximpanzés, no a nivell de país o regió sinó a nivell molt més local, per exemple de parc nacional. Per aconseguir-ho caldran moltes més mostres de les quals en coneguem el seu origen geogràfic i és aquí on les mostres no-invasives ens seran de gran ajuda. Espero poder-vos explicar una mica més sobre aquest tema en un futur ben pròxim!
Frandsen et al. Targeted conservation genetics of the endangered chimpanzee. Heredity 2020. DOI: https://doi.org/10.1038/s41437-020-0313-0