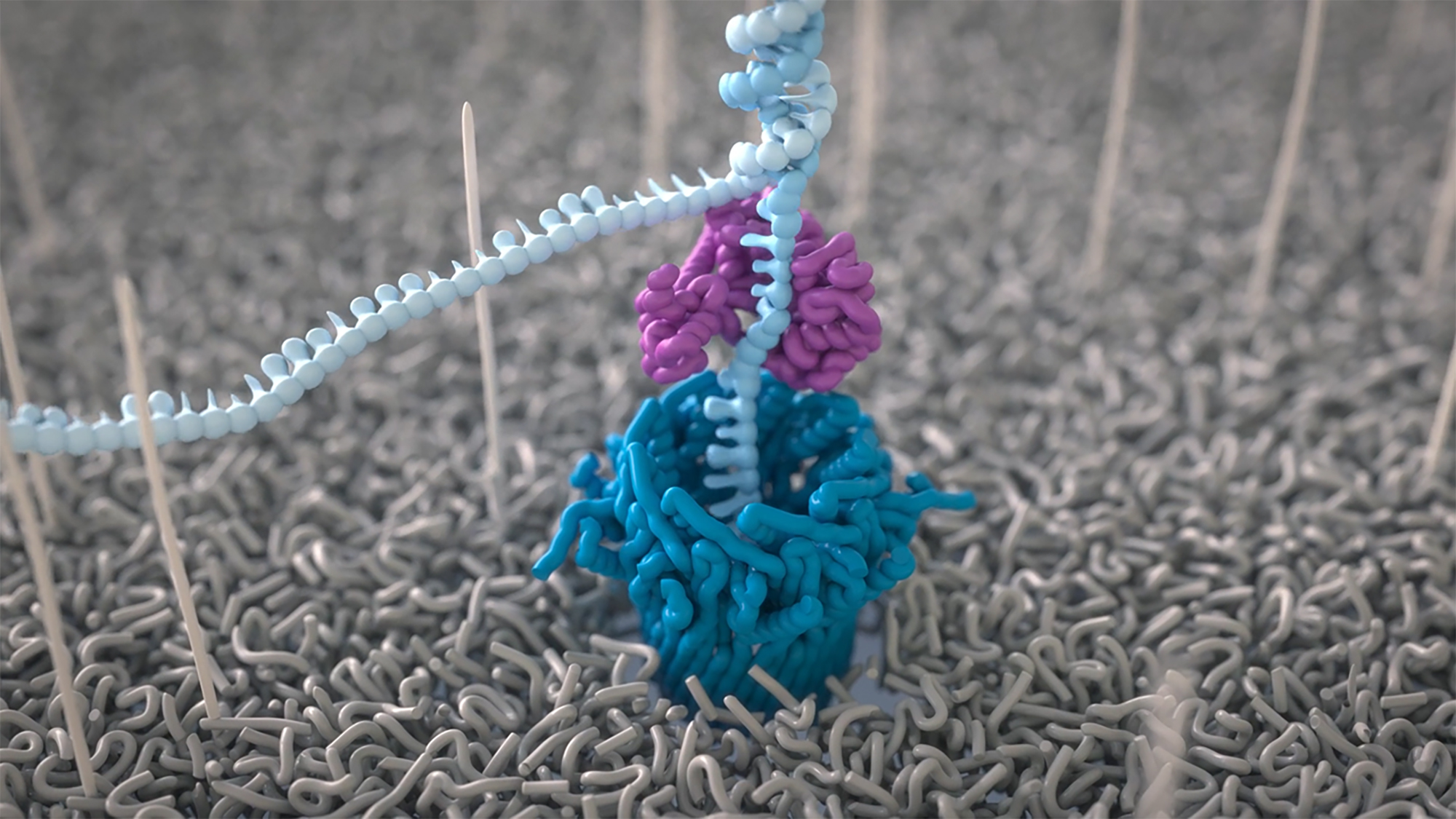
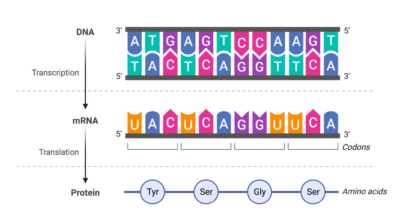
Un equipo científico del Centro de Regulación Genómica (CRG) ha publicado un nuevo método, basado en algoritmos computacionales y deep learning, que permite la detección de ARN en tiempo real analizando múltiples muestras simultáneamente.
El método se basa en la secuenciación de nanoporos, una técnica que proporciona información inmediata de ADN y ARN de una célula viva. La desventaja de la secuenciación de nanoporos, cuando se aplica a muestras de pacientes, es que requiere una gran cantidad de material biológico. El nuevo método desarrollado por el equipo de Eva M. Novoa permite identificar ARN de muchas muestras diferentes secuenciadas al mismo tiempo, disminuyendo la necesidad de material biológico de entrada de cada paciente y el coste de la técnica.
El algoritmo identifica y clasifica moléculas de ARN y permite obtener bibliotecas de ARN de alta calidad mediante un código de barras de cada muestra con un 99% de precisión.
“La secuenciación directa de ARN proporciona información compleja e imparcial, como los virus presentes en una muestra, su proporción o las mutaciones presentes en cada virus. Una información mucho más rica que la simple presencia / ausencia de un virus específico proporcionada por una PCR ”
Eva Novoa, líder de grupo del CRG
El método abre la posibilidad de aplicar esta tecnología a muestras derivadas de pacientes y presenta una alternativa más rápida y rica a las formas actuales de detección del SARS-CoV-2.
Smith M, Ersavas T, Ferguson J, Liu H, Lucas M, Begilk O, Bojarski L, Barton K, Novoa, E. 2020. «Molecular barcoding of native RNAs using nanopore sequencing and deep learning«. Genome Research, doi: https://doi.org/10.1101/gr.260836.120